GBlocks: Aplicaciones en la Biología Sintética
Los GBlocks han revolucionado la Biología Sintética al ser un parteaguas en la síntesis de ADN de doble cadena. A diferencia de la secuenciación de nueva generación, la síntesis del ADN no ha seguido la misma tendencia de disminución de costos y alto rendimiento. Sin embargo, en 2013 IDT desarrollo los Gblocks: moléculas de ADN de doble cadena con una longitud desde 125 pares de bases (pb) hasta 3000 pb, con una tasa de error media menor a 1:5000 pb y, con una alta calidad debido a los procesos de verificación a los que se someten.
¿Cómo estamos seguros que las secuencias se conservan en la síntesis?
Todos los GBlocks pasan por un control de calidad de 3 pasos:
- Electroforesis capilar: Verifica que la longitud de la secuencia sea la correcta.
- Espectrometría de Masas: Verifica que la masa de los Gblocks sea la correcta.
- Secuenciación: Verifica el orden de los nucleótidos en la secuencia.
Sin embargo, hay otros fragmentos más complejos que están sujetos a proceso de calidad más estrictos como Next Generation Sequencing (NGS). Este es el caso de los GBlocks HiFi Gene Fragments.
Los GBlocks HiFi Gene Fragments son fragmentos de doble cadena de ADN de entre 100 pb hasta 300 pb que son verificados en calidad por NGS, y por consiguiente, la tasa de error disminuye aún más: 1:12,000. Estos fragmentos de alta calidad permiten el ensamblaje de genes facilitando el proceso de clonación en el cual se requiere fidelidad, reduciendo la inserción o sustitución de bases, y flexibilidad, al poder clonarse en cualquier vector de manera práctica.
Por si fuera poco, los GBlocks resultan aún más prácticos cuando la secuencia que utilizaremos en nuestras aplicaciones requiera variaciones en una región. Los GBlocks Gene Fragment Libraries son GBlocks en pool o agrupados de 251 pb a 500 pb que resultan muy útiles en el estudio de anticuerpos recombinantes para ingeniería de proteínas. Las regiones variables pueden ser hasta 18 pb consecutivas N (cualquier base; A, G, C o T) y K (G o T) localizados al menos a 125 pb de cada extremo del fragmento (Figura 1).

Figura 1. Representación de GBlocks Gene Fragment Libraries con una región variable de hasta 18 pb ubicados al menos a 125 pb de cada extremo de los fragmentos.
Sin embargo, las aplicaciones van más allá de construcciones prácticas y estudios de anticuerpos recombinantes. Actualmente, los GBlocks están citados en más de 8 millones de publicadas reportadas en la base de datos de NCBI con una gama amplia de aplicaciones como edición genómica basada en CRISPR-CAS9, y controles de qPCR y NGS, que revisaremos a continuación (Figura 2).
Anticuerpos Recombinantes
 Los GBlocks resultan particularmente útiles en el estudio de anticuerpos recombinantes en donde se requiere evaluar la eficacia de distintas secuencias de anticuerpos sobreexpresados, generados en pacientes después de una inmunización a un patógeno. Tal es el caso de la publicación de Chen. Et. al. 2021, en la cual proponen un protocolo para la selección basada en phage display de dominios de anticuerpos VH neutralizantes de Sars-CoV-2. Las cadenas largas de los anticuerpos se construyen con GBlocks en vez de realizar la purificación de productos de PCR.
Los GBlocks resultan particularmente útiles en el estudio de anticuerpos recombinantes en donde se requiere evaluar la eficacia de distintas secuencias de anticuerpos sobreexpresados, generados en pacientes después de una inmunización a un patógeno. Tal es el caso de la publicación de Chen. Et. al. 2021, en la cual proponen un protocolo para la selección basada en phage display de dominios de anticuerpos VH neutralizantes de Sars-CoV-2. Las cadenas largas de los anticuerpos se construyen con GBlocks en vez de realizar la purificación de productos de PCR.
Estudio de regiones genómicas
Al estudiar regiones variables, también resulta conveniente utilizar GBlocks para evaluar secuencias de interacción o regulatorias en el genoma o entre transcritos, por ejemplo:
 Interacciones de ADN-proteínas como las uniones de factores de transcripción a promotores.
Interacciones de ADN-proteínas como las uniones de factores de transcripción a promotores.- Interacciones ADN-ARN que forman estructuras como los tríplex de ADN-ARN
- Eficacia de promotores
Por ejemplo, en un estudio evaluaron la eficacia de diferentes promotores sintéticos derivados de pararetrovirus con la finalidad de generar promotores eficientes y universales para plantas mono y dicotiledóneas. Para logarlo, se sintetizaron promotores quiméricos como GBlocks a partir de secuencias de promotores prexistentes y posteriormente evaluaron su eficacia clonando un gen reportero bajo los promotores sintéticos (Gupta, D. et. Al. 2021).
Construcciones
 El realizar construcciones es un trabajo complejo y puede requerir varios días. Sin embargo, los GBlocks llegaron a facilitar este proceso. Debido a su característica de ser moléculas de ADN de doble cadena, liofilizados y con una longitud de hasta 3000 pb, fácilmente podemos sustituir la amplificación de fragmentos múltiples por PCR y su posterior ligación por un solo fragmento sintético de Gblock, listo para clonarse en cualquier vector gracias a que pueden ordenarse con los extremos fosforilados.
El realizar construcciones es un trabajo complejo y puede requerir varios días. Sin embargo, los GBlocks llegaron a facilitar este proceso. Debido a su característica de ser moléculas de ADN de doble cadena, liofilizados y con una longitud de hasta 3000 pb, fácilmente podemos sustituir la amplificación de fragmentos múltiples por PCR y su posterior ligación por un solo fragmento sintético de Gblock, listo para clonarse en cualquier vector gracias a que pueden ordenarse con los extremos fosforilados.
Un ejemplo de su aplicación al realizar construcciones es al realizar la clonación de fragmentos en vectores para evaluar interacciones en sistemas reporteros, para lo cual siempre hay que clonar una versión wild type y una mutante.
Al evaluar interacciones microRNAs y sus potenciales blancos, requerimos de estos sistemas de vectores reporteros. En este caso los GBlocks se utilizan para generar las construcciones de varios potenciales sitios de interacción en su versión wild type y mutante. Por ejemplo, en esta publicación evaluaron la regulación de dos microRNAs de virus del ébola; miR-MAY-251 and miR-MAK-403 para determinar la regulación sobre potenciales mRNAs blancos humanos involucrados en el fenotipo hemorrágico de la enfermedad, respuesta inmunitaria y modulación de la replicación del virus. En este caso los sitios de interacción fueron construidos por Gblocks (Diallo, I. et. al. 2022).
Edición Genómica
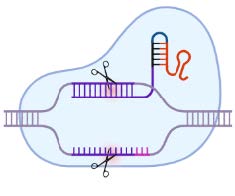 El sistema de Edición Genómica basado en el mecanismo de CRISPR CAS9 requiere de tres componentes: gRNA + ADN donador + CAS9. Estos tres componentes son ingresados a la célula mediante varios métodos, uno de ellos es utilizando vectores. El gRNA tiene un papel clave en guiar a la proteína CAS9 a su blanco genómica para realizar la edición. Debido a que los gRNAs pueden tener off-targets y variar en eficiencia según la ubicación de hibridación, se diseñan más de un gRNA por región a editar. En este contexto resulta muy práctico partir de GBlocks para ingresar varios gRNAs a los vectores.
El sistema de Edición Genómica basado en el mecanismo de CRISPR CAS9 requiere de tres componentes: gRNA + ADN donador + CAS9. Estos tres componentes son ingresados a la célula mediante varios métodos, uno de ellos es utilizando vectores. El gRNA tiene un papel clave en guiar a la proteína CAS9 a su blanco genómica para realizar la edición. Debido a que los gRNAs pueden tener off-targets y variar en eficiencia según la ubicación de hibridación, se diseñan más de un gRNA por región a editar. En este contexto resulta muy práctico partir de GBlocks para ingresar varios gRNAs a los vectores.
Un ejemplo de esta aplicación, es una terapia de edición genética para tratar el Síndrome Reproductivo y Respiratorio del Cerdo (PRRS). En esta terapia generaron embriones de cerdo CD163 nulos, el receptor del virus causante del síndrome. Para ello utilizaron como gRNA; GBlocks. El resultado fue que las crías CD163 eran inmunes a una infección causada por el virus del PRRS (Whitworth, K.M. and Prather, S.R. 2017).
.png)